Bernhard
Peter
Der
natürliche Ligand der Neuraminidase: Sialinsäure
Die folgenden Abbildungen zeigen, wie die Neuraminidase funktioniert: In einer tiefen Tasche ist die Bindungsstelle für Sialinsäure, das aktive Zentrum. Wenn nun ein Rezeptor mit Sialinsäure von der Zelloberfläche mit der Neuraminidase in Kontakt kommt, gleitet die Sialinsäure in das aktive Zentrum, worauf das Enzym hydrolytisch tätig wird und die Bindung spaltet. Dieser Mechanismus ist Voraussetzung dafür, daß sich dir frisch produzierten und freigesetzten Influenza-Viren von der Zelle ablösen können.

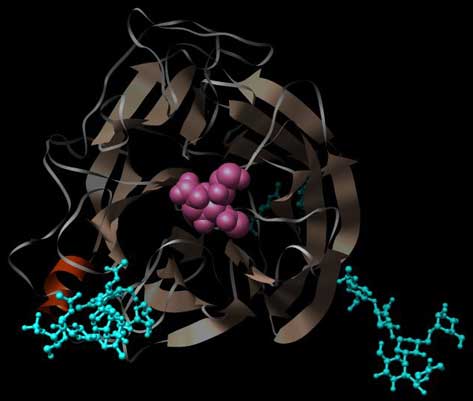
Abb.: Ansicht einer monomeren Neuraminidase, deren Proteinteil als Sekundärstruktur und deren Zuckerreste als Kugel-Stab-Modell dargestellt sind. Im aktiven Zentrum in der Mitte befindet sich ein Molekül Sialinsäure als Ligand. PDB-ID: 2BAT.
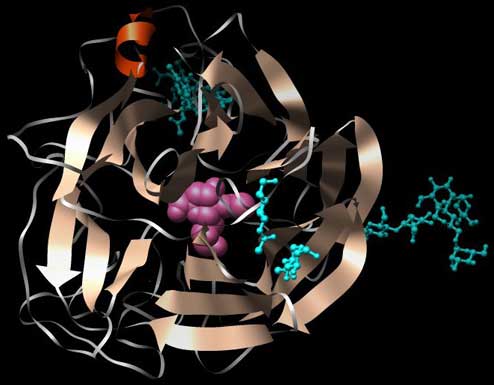
Abb.: Wie oben, Ansicht von der Rückseite. PDB-ID: 2BAT.
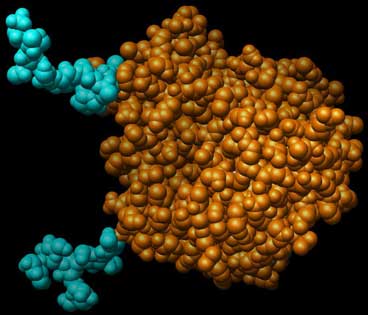
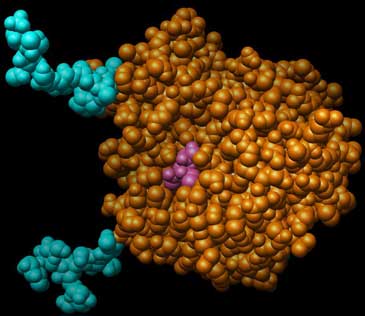
Abb.: Ansicht einer monomeren Neuraminidase, deren Proteinteil und und deren Zuckerreste sphärisch dargestellt sind. Links Darstellung ohne Ligand, rechts mit einem Molekül Sialinsäure als Ligand. PDB-ID: 2BAT.
Grundlage für obige Abbildungen: Research
Collaboratory for Structural Bioinformatics Protein Data Bank
http://www.rcsb.org/pdb, PDB-ID: 2BAT Varghese, J. N., Colman, P.
M. THE STRUCTURE OF THE COMPLEX BETWEEN INFLUENZA VIRUS
NEURAMINIDASE AND SIALIC ACID, THE VIRAL RECEPTOR,
Originalbeitrag: Varghese, J. N., McKimm-Breschkin, J. L.,
Caldwell, J. B., Kortt, A. A., Colman, P. M. ![]() The structure of the
complex between influenza virus neuraminidase and sialic acid,
the viral receptor. Proteins v14 S. 327-332, 1992
The structure of the
complex between influenza virus neuraminidase and sialic acid,
the viral receptor. Proteins v14 S. 327-332, 1992
Wie erwähnt, findet sich die Neuraminidase zu Vierer-Assoziaten auf der Oberfläche zusammen. Die folgenden Abbildungen tragen dieser Tatsache Rechnung und zeigen eine komplette Vierer-Einheit. Es handelt sich um eine Neuraminidase des Subtyps N6, gefunden in Enten, die ein sehr interessantes Detail aufweist: Sie hat nämlich in jeder Untereinheit zwei Bindungsstellen für Sialinsäure. Dabei wird eine Sialinsäure in einer relativ tiefen Tasche, die andere mehr oberflächlich gebunden.
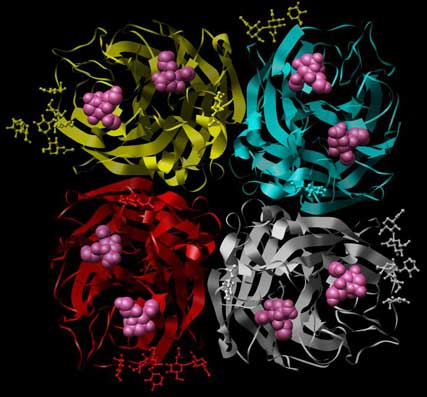
Abb.: Ansicht einer tetrameren Neuraminidase, deren Proteinteil jeweils als Sekundärstruktur und deren Zuckerreste als Kugel-Stab-Modell dargestellt sind, jede Untereinheit in einer anderen Farbe. Der Ligand Sialinsäure ist als sphärisches Modell in magenta dargestellt. PDB-ID: 1W20.
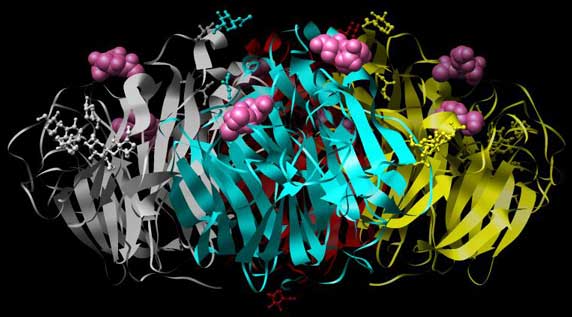
Abb.: Wie oben, Seitenansicht. PDB-ID: 1W20.
Grundlage für obige Abbildungen: Research Collaboratory for Structural Bioinformatics Protein Data Bank http://www.rcsb.org/pdb, PDB-ID: 1W20 Rudino-Pinera, E., Tunnah, P., Crennell, S. J., Webster, R. G., Laver, W. G., Garman, E. F. STRUCTURE OF NEURAMINIDASE FROM ENGLISH DUCK SUBTYPE N6 COMPLEXED WITH 30 MM SIALIC ACID (NANA, NEU5AC), CRYSTAL SOAKED FOR 3 HOURS AT 291 K, Originalbeitrag: Rudino-Pinera, E., Tunnah, P., Crennell, S. J., Webster, R. G., Laver, W. G., Garman, E. F. The Crystal Structure of Type A Influenza Virus Neuraminidase of the N6 Subtype Reveals the Existence of Two Separate Neu5Ac Binding Sites, in Veröffentlichung.
Das Enzym der Freiheit: Neuraminidase - Der Mechanismus der Vermehrung - Grippe-Medikamente - Neuraminidase-Hemmer - Literatur, Quellen und Links
Zurück zur Übersicht Grippe /
Influenza
Andere pharmazeutische Seiten
![]() Home
Home
©
Copyright Text, Graphik und Photos: Bernhard Peter 2006
Impressum