Bernhard
Peter
So
setzt man die Reverse Transkriptase außer Gefecht: NNRTI -
nicht-nukleosidische Hemmstoffe
Im Gegensatz zu den Antimetaboliten, die der Reversen Transkriptase wie ein Kuckucksei untergeschoben werden und Nukleotid spielen, worauf die Kettenverlängerung unterbrochen wird, funktionieren die NNRTIs anders. Sie haben chemisch keine Ähnlichkeit mit Substraten der Polymerase. Die Polymerase-Einheit der Reversen Transkriptase versucht auch nicht, sie in den gerade bearbeiteten Strang einzubauen. Aber die Arzneistoffe besitzen eine Struktur, die eine hohe Bindungsaffinität zu einer anderen Stelle der Reversen Transkriptase hat. Auf der Rückseite des aktiven Zentrums tritt die Substanz Nevirapin beispielsweise in Wechselwirkung mit der Eiweißstruktur und bindet an einem sog. allosterischen Zentrum. Durch die Bindung verändert sich die Struktur des Enzyms ein klein wenig. Das reicht aber schon aus, daß das aktive Zentrum eine andere Form hat als vorher und nicht mehr richtig funktioniert. Man spricht von einer allosterischen Hemmung, einer Hemmung der Reversen Transkriptase durch Bindung an einer anderen als der aktiven Stelle.

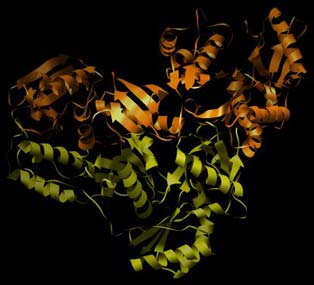
Abbildung der Reversen Transkriptase von HIV-1 ohne Ligand (Protein Data Base PDB-ID: 1DLO). Zur besseren Übersichtlichkeit ist hier nur die Sekundärstruktur (Helix und Faltblatt) der RTase wiedergegeben. Deutlich zu erkennen ist hier die Zusammensetzung aus zwei Ketten und Aufteilung in zwei Funktionalitäten: Orange ist die Polymerase, gelb ist die Nuklease. Die RNA/DNA bindet im oberen Teil zur Ergänzung mit weiteren Nukleotiden. Genau diese hier in orange eingefärte Polymerase-Funktionalität ist das Ziel der NNRT-Hemmstoffewie z. B. Delavirdin, Efavirenz, Nevirapin.
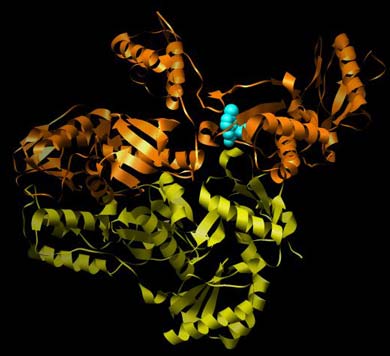
Abbildung der Reversen Transkriptase von HIV-1 mit dem nicht-kompetitiven Hemmstoff Efavirenz (Protein Data Base PDB-ID: 1FK9). Zur besseren Übersichtlichkeit ist hier nur die Sekundärstruktur (Helix und Faltblatt) der RTase wiedergegeben. Durch die enge Wechselwirkung mit dem Hemmstoff Efavirenz kommt es zu einer Änderung der Raumstruktur, die das Funktionieren des Enzyms beeinträchtigt.
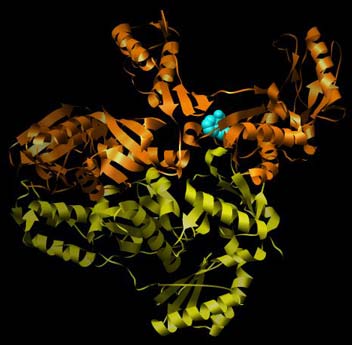
Abbildung der Reversen Transkriptase von HIV-1 mit dem nicht-kompetitiven Hemmstoff Nevirapin (Protein Data Base PDB-ID: 1FK9). Zur besseren Übersichtlichkeit ist hier nur die Sekundärstruktur (Helix und Faltblatt) der RTase wiedergegeben. Durch die enge Wechselwirkung mit dem Hemmstoff Nevirapin kommt es zu einer Änderung der Raumstruktur, die das Funktionieren des Enzyms beeinträchtigt.
Wer genau hinsieht, wird erkennen, daß die Sekundärstruktur der Reversen Transkriptase leichte Abweichungen zwischen den einzelnen Bildern zeigt. Das liegt daran, daß durch die Wechselwirkung zwischen Enzym und Hemmstoff leicht veränderte Raumstrukturen eingenommen werden. Und genau das ist der Sinn der Sache!
Literatur:
Research Collaboratory for
Structural Bioinformatics Protein Data Bank
http://www.rcsb.org/pdb, PDB-ID: 1JLB, CRYSTAL STRUCTURE OF Y181C
MUTANT HIV-1 REVERSE TRANSCRIPTASE IN COMPLEX WITH NEVIRAPINE,
Ren, J., Nichols, C., Bird, L., Chamberlain,
P., Weaver, K., Short, S., Stuart,
D.I., Stammers, D.K. Structural mechanisms of drug
resistance for mutations at codons 181 and 188 in HIV-1 reverse
transcriptase and the improved resilience of second generation
non-nucleoside inhibitors. J.Mol.Biol. v312 pp.795-805, 2001
Research Collaboratory for Structural Bioinformatics Protein Data
Bank http://www.rcsb.org/pdb, PDB-ID: 1DLO, HUMAN
IMMUNODEFICIENCY VIRUS TYPE 1, Reverse Trankriptase ohne Ligand,
Hsiou, Y., Ding, J., Das,
K., Clark Jr., A.D., Hughes,
S.H., Arnold, E. Structure of unliganded HIV-1 reverse
transcriptase at 2.7 A resolution: implications of conformational
changes for polymerization and inhibition mechanisms. Structure
v4 pp.853-860, 1996
Research Collaboratory for Structural Bioinformatics Protein Data
Bank http://www.rcsb.org/pdb, PDB-ID: 1FK9, CRYSTAL STRUCTURE OF
HIV-1 REVERSE TRANSCRIPTASE IN COMPLEX WITH DMP-266 (EFAVIRENZ),
Ren, J., Milton, J., Weaver,
K.L., Short, S.A., Stuart,
D.I., Stammers, D.K. Structural basis for the
resilience of efavirenz (DMP-266) to drug resistance mutations in
HIV-1 reverse transcriptase. STRUCTURE FOLD.DES. v8 pp.1089-1094
, 2000
So
funktionieren HIV-Medikamente:
Ein Schlüsselenzym: Die Reverse
Transkriptase
So setzt man die Reverse Transkriptase
außer Gefecht: Antimetabolite
So setzt man die Reverse Transkriptase
außer Gefecht: nicht-nukleosidische Hemmstoffe
Schlüsselenzym Protease: Die chemische
Schere
Wir setzen die chemische Schere außer
Betrieb: Protease-Hemmstoffe
NNRTI:
Nicht-nukleosidische Reverse-Transkriptase-Hemmer
Delavirdin, Efavirenz, Nevirapin
Zurück zur Übersicht HIV / AIDS
Andere pharmazeutische Seiten
![]() Home
Home
©
Copyright Text, Graphik und Photos: Bernhard Peter 2006
Impressum